水环境eDNA检测
技术介绍
环境DNA(Environmental DNA,eDNA),即环境样本中生物体的DNA痕迹。在各种环境包括土壤、沉积物、排泄物、空气、水体中,生活着各种各样的生物体,这些生物个体的生命活动会将自身的DNA掉落到环境中,这些释放到环境中的生物体DNA就是环境DNA。
水环境eDNA检测技术主要依托于对水体样本中的DNA片段的检测来实现对水体中生活的相关物种的存在检测。通过对水体中eDNA的提取、高通量测序及序列比对流程,即可获取环境样本中DNA所属物种的分类学信息,进而研究水环境中各物种多样性程度、生活史过程、濒危程度等。希望透过此技术揭开自然界生态之密,并赋能保护生物多样性为首要目标。
技术优势
采用简单:仅需采集水样即可进行鱼类、藻类、底栖动物、微生物等多层次的物种多样性分析。
生态无损伤:不捕捞实体生物,对调查地生态系统的扰动较少。
不依赖经验:对物种的鉴定通过物种DNA数据库完成,不依赖鉴定人员的经验。
灵敏度高:检出灵敏度高,即便是丰度很低的稀有物种也可以检测,适用于对珍稀物种或外源新物种入侵的监测。

eDNA检测技术通常被应用于
1、物种鉴定和物种多样性评估
2、外来入侵物种分析和物种监测
3、构建污染物与生物群落结构之间的关系
4、预测评估生态系统的健康程度
常规鉴定(形态学、生理生化特征)
微生物常规鉴定主要为形态结构、培养特征观察和特征生理生化反应等。
微生物的形态结构观察主要是通过染色,在显微镜下对其形状、大小、排列方式、细胞结构(包括细胞壁、细胞膜、细胞核、鞭毛、芽孢等)及染色特性进行观察,直观地了解细菌在形态结构上特性,根据不同微生物在形态结构上的不同达到区别、鉴定微生物的目的。
1、微生物的培养特征主要包括以下内容:
细菌的培养特征:在固体培养基上,观察菌落大小、形态、颜色(色素是水溶性还是脂溶性)、光泽度、透明度、质地、隆起形状、边缘特征及迁移性等。在液体培养中的表面生长情况(菌膜、环)混浊度及沉淀等。半固体培养基穿刺接种观察运动、扩散情况。
霉菌/酵母菌的培养特征:大多数酵母菌没有丝状体,在固体培养基上形成的菌落和细菌的很相似,只是比细菌菌落大且厚。液体培养也和细菌相似,有均匀生长、沉淀或在液面形成菌膜。霉菌有分支的丝状体,菌丝粗长,在条件适宜的培养基里,菌丝无限伸长沿培养基表面蔓延。霉菌的基内菌丝、气生菌丝和孢子丝都常带有不同颜色,因而菌落边缘和中心,正面和背面颜色常常不同,如青霉菌:孢子青绿色,气生菌丝无色,基内菌丝褐色。霉菌在固体培养表面形成絮状、绒毛状和蜘蛛网状菌落。
2、微生物的特征生理生化反应:
微生物的生理生化特性包括营养类型、碳氮源利用能力、各种代谢反应、酶活性反应、碳水化合物利用试验、氧化还原反应和血清学反应等。
在微生物检验中常用的生理生化反应有:糖酵解试验、淀粉水解试验、V-P试验、甲基红(Methyl Red)试验、靛基质(Imdole)试验、硝酸盐(Nitrate)还原试验、明胶(Gelatin)液化试验、尿素酶(Urease)试验、氧化酶(Oxidase)试验、硫化氢(H2S)试验、三糖铁(TSI)琼脂试验、硫化氢-靛基质-动力(SIM)琼脂试验等

分子生物学鉴定(细菌16SrDNA和真菌ITS鉴定)
采用比对DNA特征序列相似性的方法实现细菌和真菌的鉴定,常用于鉴定细菌和真菌的Marker基因主要有细菌16S rRNA 基因序列、真菌18S rRNA/LSU D1-D2/ITS1-5.8S-ITS2序列。
16S rRNA 基因是所有的细菌都具有的基因,长度适中(在1500~1600bp),进化上具有良好的时钟性质,结构与功能上具有高度的保守性,能很好体现不同菌属之间的差异,又能轻松利用测序技术得到其序列,故被细菌学家和分类学家普遍认可,素有“细菌化石”之称。
16S rRNA基因序列由9个可变区和10个保守区(下图一)构成。
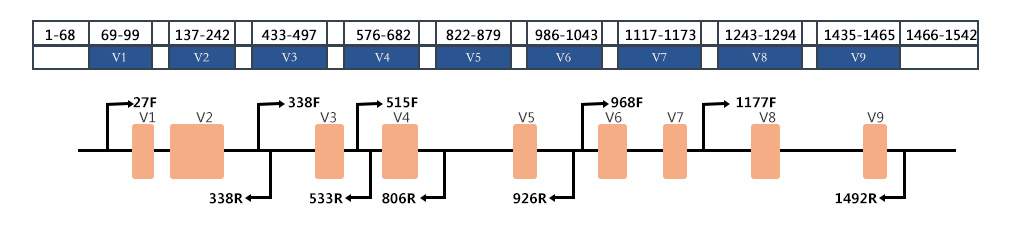
保守区序列反映了物种间的亲缘关系,可变区序列则能体现物种间的差异。通过在保守区设计引物,利用PCR反应扩增出高变区序列,然后通过一代测序技术得到高变区序列信息,再将其与数据库中已经存在的菌种对应区间的序列信息进行比对分析,来进行菌种鉴定。
基于ITS基因测序的优点
为了解决D2基因真菌鉴定的局限性,相关研究发现核糖体操纵子中的ITS序列可更好的被应用于真菌的系统鉴定。在真菌的核糖体操纵子中有两个ITS区域。ITS1位于18S和5.8S之间,ITS2位于5.8S和28S之间。整个rRNA操纵子一起转录,但转录后的rRNA,两个不具备功能的ITS被剪切掉。ITS序列作为一个间隔序列十分重要,但同时它又不具备功能,因此,它的突变速度会大于5.8S、18S和28S基因。这少量的突变提升,使得ITS的序列相比D2具备更好的多态性。因此,将ITS1-5.8S-ITS2整个测序有助于真菌分类。
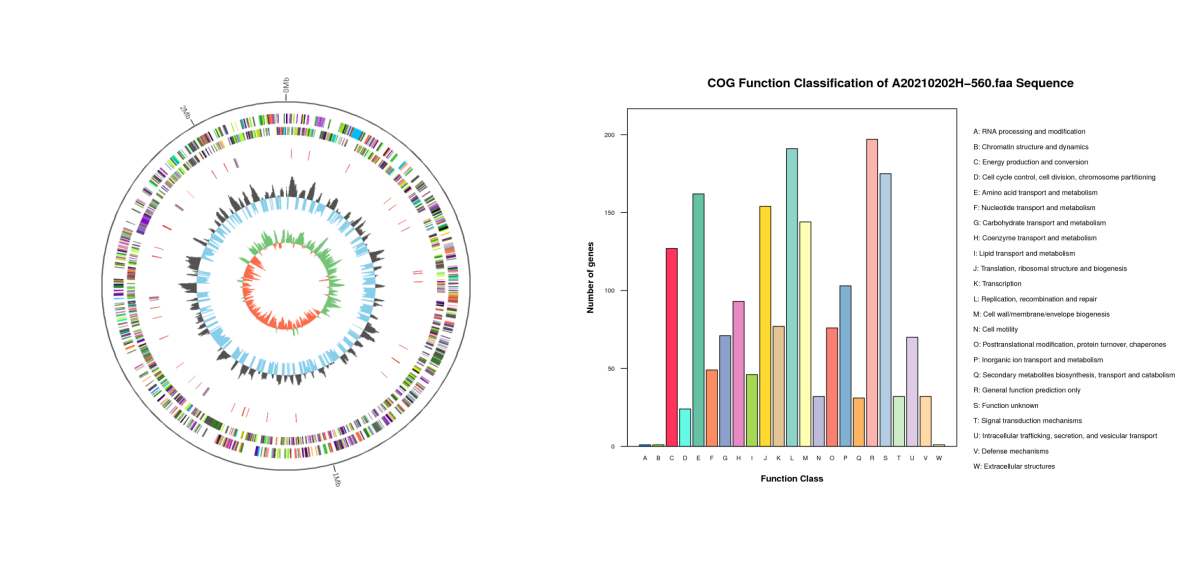
细菌/真菌基因组测序
微生物广泛存在于自然界中,与人类的生产和生活息息相关。微生物生物多样性丰富,基因组具有相对较小、重复序列较高、易于突变等特点,因此很必要对微生物进行全基因组测序,而且同动植物相比较在时间和成本上也容易实现。目前,全基因组测序成为微生物遗传进化、疾病预防控制、生物工程技术等方面重要的研究和开发手段。众所周知,基因组测序最主要的难度在于基因组的重复序列和GC偏移。微生物基因组测序的完成图(完成图要求完整基因组序列,单碱基错误率低于十万分之一,gap数不超过5个)主要有两条技术路线:第一条是以二代测序(Matepair测序文库,插入片段3Kb)配合一代测序完成,一代测序主要用于gap的填补;第二条是以三代测序为主配合二代测序。第一条技术路线往往会GC偏移的影响,同时3Kb插入片段有时不够横跨重复区域而导致测序结果的不理想,从而增加了测序费用和延长了测序时间。
1. 基因组组分分析:编码基因预测和注释、功能分类;重复序列预测;非编码RNA注释,包括tRNA,rRNA,sRNA等;
2. 基因岛预测:行使相同功能的基因聚集成簇,包括毒力岛、抗性岛、抗生素岛等;
3. 前噬菌体:可整合进基因组的噬菌体序列,常会带来功能基因;
4. CRISPR:与细菌免疫相关,抵御外来DNA序列;
5. 假基因分析:一般是进化过程中失活的基因冗余拷贝;
6. 进化分析。
应用场景
1.对于新/未知物种的判断鉴定。
2.探讨基因组中有无目标序列或基因。
3.进行多样本宏观分析,如系统进化分析、溯源分析等。
4.可在基因层面对多个样本进行比较分析,也可在局部范围内进行样本间结构的比较。
5.基础研究,挖掘基因组中的基因资源、作用机制、重要元件等,可满足绝大部分的研究需求(二代+三代)
原核(细菌)/真核(动、植物、真菌)转录组测序
转录组(transcriptome)广义上指某一生理条件下,细胞内所有转录产物的集合,包括信使RNA、核糖体RNA、转运RNA及非编码RNA;狭义上指所有mRNA的集合。蛋白质是行使细胞功能的主要承担者,蛋白质组是细胞功能和状态的最直接描述,转录组成为研究基因表达的主要手段,转录组是连接基因组遗传信息与生物功能的蛋白质组的必然纽带。
细菌转录组测序是在实验中通过去除总RNA的核糖体后进行转录组测序,该实验除了发现基因的差异表达之外,还可以对物种的基因组进行重新的注释,包括鉴定新基因,sRNA(还包括Ribo-switch),anti-sense RNA,5’UTR,4’UTR,operon等。
1. 测序质量评估分析;
2. 测序结果的随机性分析
3. RNA-Seq生物信息学分析
4. 基因表达及差异分析
5. 差异基因COG/KO/GO富集分析
6. sRNA预测及差异分析
7. UTR预测
8. Operons预测
应用场景(真核生物和原核生物检测方法有差异):
(1)了解样本基因表达情况。
(2)比较样本之间基因表达水平差异情况。
(3)了解GO和KEGG功能富集情况。
(4)了解样本SNP、InDel等结构水平信息。
常用于不同处理条件、不同处理时间、不同组织部位等基因表达差异研究。
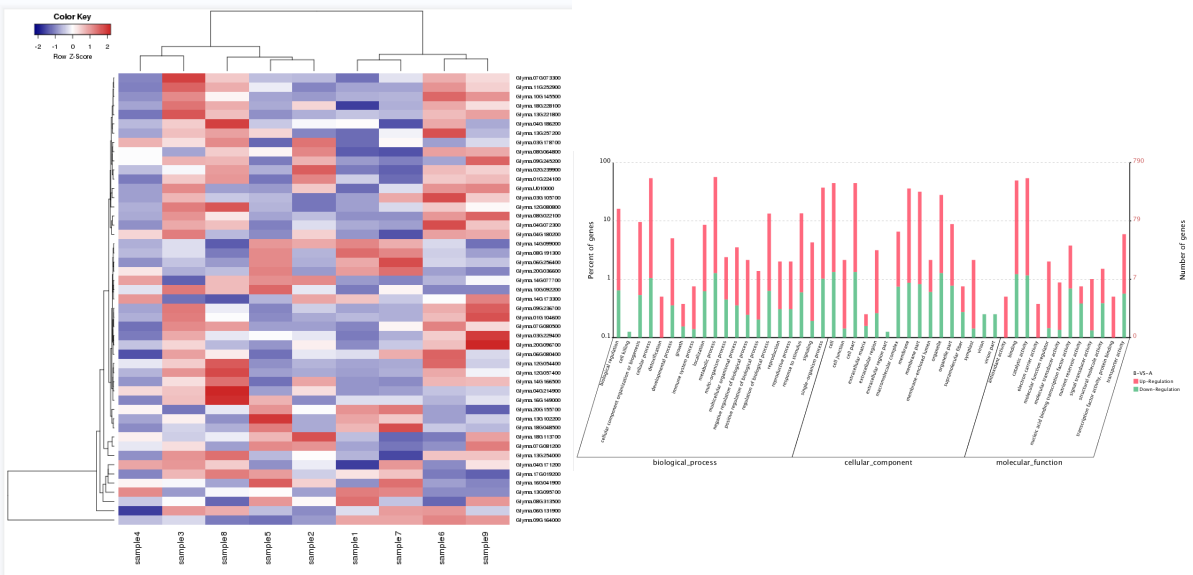
微生物宏基因组测序
宏基因组(Metagenomics),也称元基因组,利用新一代高通量测序技术(Next Generation Sequencingtechnology , NGS)以特定环境下微生物群体基因组为研究对象,在分析微生物多样性、种群结构、进化关系的基础上,进一步探究微生物群体功能活性、相互协作关系及与环境之间的关系,发掘潜在的生物学意义。与传统微生物研究方法相比,宏基因组测序技术规避了绝大部分微生物不能培养、痕量菌无法检测的缺点,因此近年来在环境微生物学研究中得到了广泛应用。
应用场景(无需分离培养,直接测序):
(1)了解目的样本微生物种类和物种丰度。
(2)了解不同样本间微生物物种组成和功能差异。
(3)了解样本中耐药、毒力等其他的功能基因。
常用于检测水体、土壤、粪便和其他存在微生物的样本

微生物多样性测序(16S/ITS扩增子测序)
16S/ITS扩增子测序是对样本中细菌的16S rRNA和真菌的ITS基因序列进行高通量测序。细菌的16S rRNA基因和真菌的ITS基因结构既具有保守性,又具有高变性。通过对某一段高变区序列(如V3-V4区域或ITS1区域)或全长进行PCR扩增后进行高通量测序,与数据库比对得到物种注释信息,可以了解样本中的菌群组成及丰度,以及组间差异菌群。
应用场景(无需分离培养,直接测序):
(1)了解样本中的物种组成。
(2)了解目的样本内物种丰富度和均匀度信息。
(3)了解样本之间的多样性关系,反映生物群落间的分化程度。
(4)用不同统计学方法分析不同样本/组间物种组成及群落组成差异,寻找具有统计学差异的biomaker。
常用于检测水体、土壤、粪便和其他存在微生物的样本。
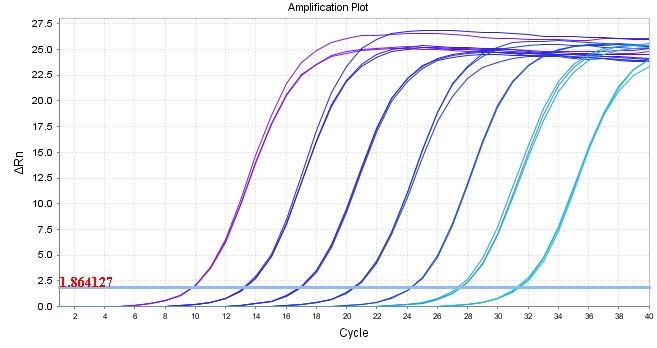
荧光定量PCR检测
实时荧光定量PCR技术,是指在PCR反应体系中加入荧光基团,利用荧光信号积累实时监测整个PCR进程,最后通过标准曲线对未知模板进行定量分析的方法。
应用场景:
(1)检测目的基因表达水平,比较目的基因之间表达水平差异。
(2)检测样本中基因拷贝数,包括目的微生物检测。
菌株选育与冻干保存服务
可根据客户要求,进行菌种筛选,包括初筛与复筛,选育具有理想性状的微生物菌株,并提供菌株的培养与保存方案。
菌种保藏方法中最有效的方法之一,适用于菌种长期保存,一般可保存数年至十余年,对一般生活力强的微生物及其孢子以及无芽胞菌都适用。在最适培养基中用最适温度培养,使培养出良好的培养物。细菌和酵母的菌龄要求超过对数生长期,一般,细菌要求24-48小时的培养物;酵母需培养3天;形成孢子的微生物则宜保存孢子;放线菌与丝状真菌则培养7-10天。




